WES 数据多类型变异分析
WES作为最为常见的临床变异检测手段,在单核苷酸突变和小片段插入缺失检测上已经有了较为成熟的流程,例如经典的GATK流程和samtools变异检测流程。但是随着数据的不断积累,尤其是对其他变异类型,如长片段的插入缺失和CNV的检测,已越来越受到生物信息研究者的重视。为此,极道科技为用户提供了目前较为常见的,通过WES或其他靶向测序数据,对长片段变异和CNV检测的工具。
1. 通过Pindel检测长片断的Indel
pindel是一款较为成熟的结构变异检测工具,其可以通过双端测序的短序列对长片断的删除(large deletions)、中等长度的插入或颠换(medium sized insertions, invertion), 串联重复(tandem duplications)和其他结构变异在单碱基水平给出突变位点。
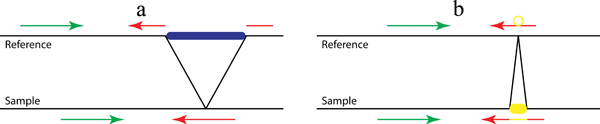
当双端测序数据(PE data)比对时,绝大多数的reads对,两端会都比对到参考基因组上。 但是小部分的只有一端会比对到参考基因组。这种情况下,没有比对至参考基因组的read,有一定的可能原因是样本的基因组中存在着长的插入或缺失,如下图所示。因此,这类没有比对上的reads,其实携带了插入缺失信息事件的信息。如果能找到一个合适的位置,将没有得到比对的一端read,进行分段后再比对回参考基因组,那么就可以得到插入或缺失发生的准确位置。当支持数足够多的时候,就可以推断出变异的发生。
2. 通过 Scalpel 给出插入缺失的具体信息
Pindel的设计初衷主要是为了发现插入缺失发生的具体位置,而对其中具体发生序列信息,没有给出具体的检测结果(至少官方给出的信息,一直强调的是变异发生位置,而不是具体插入了什么)。
Scalpel是另外一种针对外显子的插入缺失检测工具,其原理为对比对结果进行局部组装,得到对应区域的组装结果后,对变异情况进行检测。虽然局部组装思想在很多的工具中都有应用(最典型的,就是GATK的HC,其之所以速度慢很大一部分原因就是在于其很负责地进行了局部组装),但是Scalpel的优势在于其可以适应基因组上的重复区域(repeat),以及局部组装过程中的参数自动调整。
3. 通过CNVkit 检测拷贝数变异
所谓拷贝数变异,其本质也是结构变异的一种,其研究目前在癌症领域应用较为广泛。但在胚系变异检测中,尤其是遗传病领域也有着较高的应用程度。除了外显子测序之外,目前也有CNV-Seq 测序方法应用于CNV的检测。
一般而言,CNV的检测其本质就是对比对结果在基因上的分布均匀程度给出推断,如果在特定区域的覆盖深度高或低,则表示区域有可能为CNV区域。理论上对于覆盖程度应该较为均匀,但是实际上,由于测序的GC偏好程度或局部不均匀造成的偏移,或测序深度不足带来的系统性偏差,会给CNV检测带来较大的误差。
因此,目前主流的针对胚系突变的CNV检测,都需要大量数据来给出一个baseline,以帮助识别;而肿瘤成对样本(即来自同一个体的正常组织和肿瘤组织),则一般需要正常样本和肿瘤样本两个比对结果进行输入,以正常样本的比对结果作为参照,来对肿瘤样本进行检测。CNVkit是目前该类型软件中,公认度较高的应用之一。
小结
从目前的生信数据文章中,我们不难发现,对于数据的处理已经不是单纯的跑一个GATK就可以满足需求了。除了SNV和小片段Indel之外,结构变异也是目前大部分文章中列举的变异检测结果。而即便是SNV,较高水平的杂志也会报道超过一种以上的变异检测工具的结果。今后achelous社区上还会为读者介绍和提供更多的工具和内置流程。欢迎读者使用或提出宝贵意见 :)
